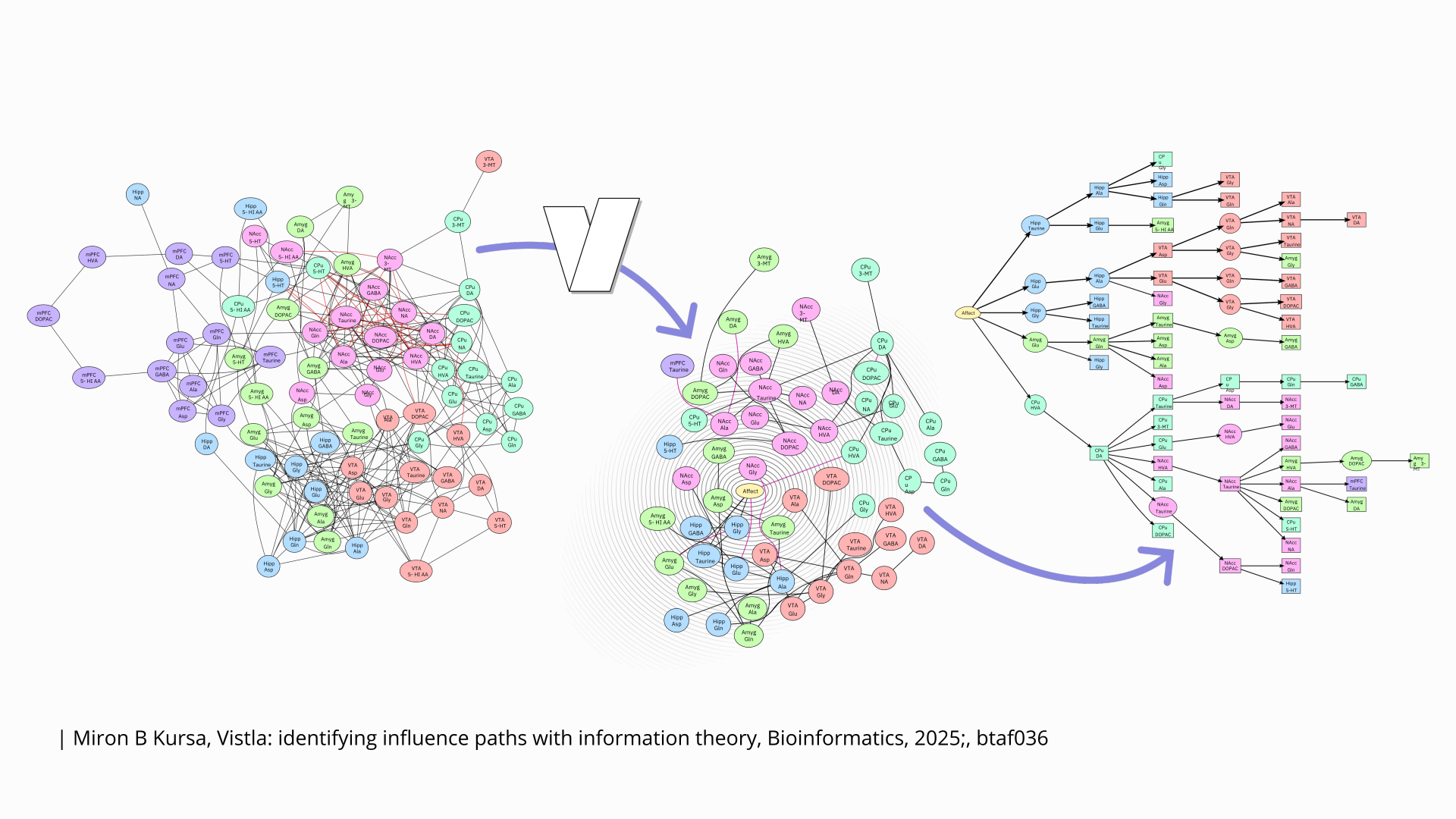
Jak rozpleść „stóg siana”?
W Bioinformatics ukazał się artykuł dr. Mirona Kursy z ICM UW pt. „Vistla: identifying influence paths with information theory„. Opisuje on algorytm Vistla służący do analizy danych z obserwacji systemów złożonych, w szczególności tych spotykanych w organizmach żywych, badanych wysokoprzepustowymi metodami molekularnymi.
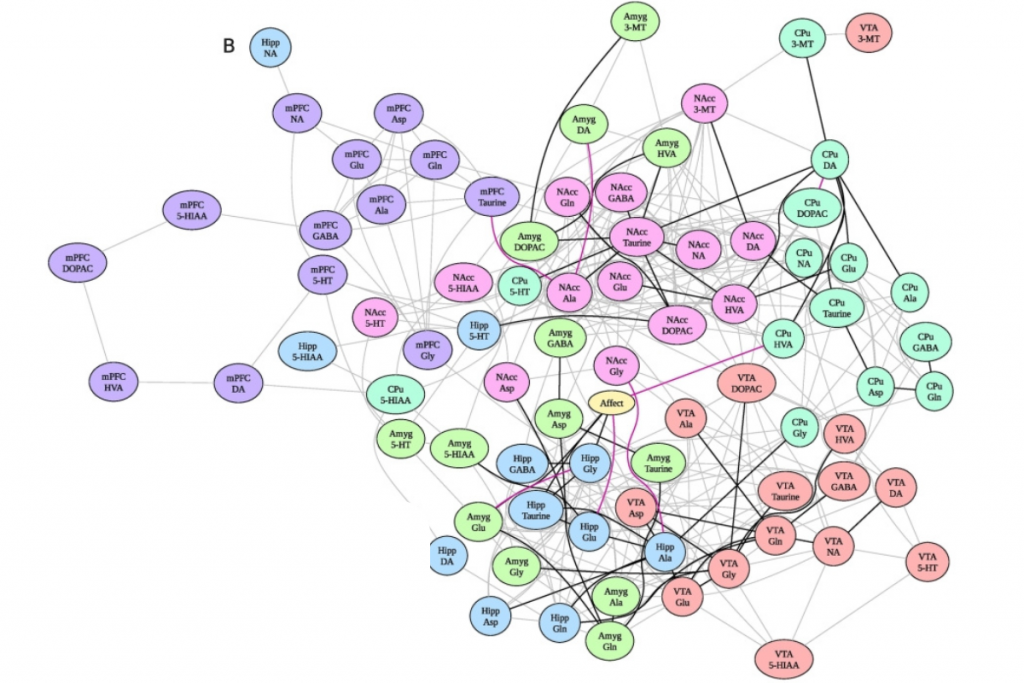
Systemem złożonym nazywamy układ, którego części składowe wchodzą w liczne, nietrywialne interakcje między sobą. Graf tychże zależności, ze względu na gęstość krawędzi i przynajmniej pozorny brak struktury, często określa się mianem „stogu siana”. Złożoność ta stanowi oczywiste wyzwanie przy próbie rozpracowania konkretnych mechanizmów działania systemu, czy też ukierunkowania jego działania na pożądane tory.
Vistla pozwala analizować dane z eksperymentów, w których system został poddany zewnętrznej interwencji, np. podaniem substancji. Metoda śledzi, jak informacja o tym zdarzeniu rozchodzi się w systemie, przechodząc między poszczególnymi komponentami – w efekcie powstaje dużo łatwiejszy w interpretacji graf o topologii drzewa.
Otwartoźródłową implementację metody dla środowiska R można znaleźć w repozytorium CRAN: https://doi.org/10.32614/CRAN.package.vistla